Krebsimmuntherapie
- Martin Döhring

- 14. Sept. 2025
- 3 Min. Lesezeit
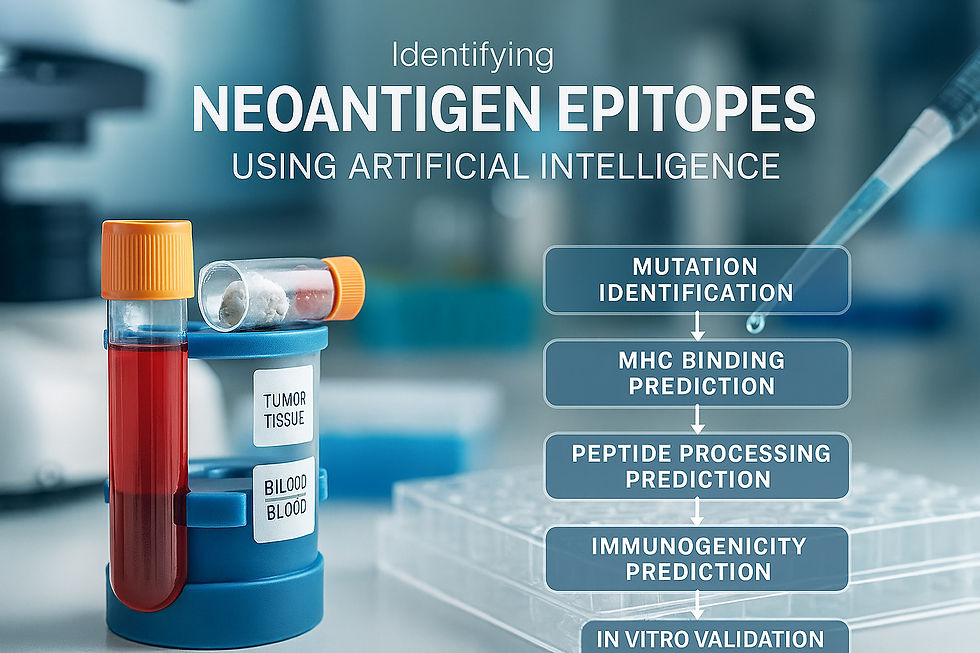
Ergänzungen zur KI-gestützten Neoantigen-Identifizierung
1. Neueste KI-Modelle und Algorithmen
Die Entwicklung von KI-Modellen für Neoantigen-Identifizierung hat in den letzten Jahren enorme Fortschritte gemacht. Neben den von dir erwähnten Tools wie NetMHC und NetMHCpan (für MHC-Bindungsvorhersage) oder NetChop (für Peptidprozessierung) gibt es mittlerweile noch leistungsfähigere Ansätze:
Transformator-basierte Modelle: Modelle wie BERT oder andere Transformer-Architekturen, die ursprünglich für die Verarbeitung natürlicher Sprache (NLP) entwickelt wurden, werden zunehmend für die Bioinformatik eingesetzt. Sie können die komplexen, kontextabhängigen Beziehungen zwischen Aminosäuresequenzen, MHC-Molekülen und T-Zell-Rezeptoren besser erfassen. Ein Beispiel ist TCR-BERT, das speziell für die Vorhersage von T-Zell-Rezeptor-Epitop-Interaktionen trainiert wurde.
Graph Neural Networks (GNNs): Diese werden verwendet, um die strukturelle Dynamik der MHC-Peptid-Bindung oder TCR-Interaktionen zu modellieren. GNNs berücksichtigen die räumliche Struktur der Moleküle, was die Genauigkeit der Bindungsvorhersagen verbessert.
Multimodale KI-Modelle: Diese kombinieren verschiedene Datentypen (z. B. DNA/RNA-Sequenzen, Proteomik-Daten, immunologische Daten) in einem Modell, um präzisere Vorhersagen zu treffen. Solche Modelle nutzen oft Transfer Learning, um von großen, allgemeinen biologischen Datensätzen auf spezifische, patientenbezogene Daten zu generalisieren.
2. Integration von Multi-Omics-Daten
Ein Trend in der Neoantigen-Identifizierung ist die Integration von Multi-Omics-Daten (Genomik, Transkriptomik, Proteomik, Epigenomik). KI-Modelle können jetzt beispielsweise RNA-Expressionsdaten nutzen, um sicherzustellen, dass ein vorhergesagtes Neoantigen nicht nur theoretisch existiert, sondern auch tatsächlich exprimiert wird. Tools wie MuPeXI oder pVAC-Seq integrieren solche Daten, um die Wahrscheinlichkeit zu erhöhen, dass ein vorhergesagtes Neoantigen tatsächlich auf der Tumorzelloberfläche präsentiert wird.
3. Fortschritte in der Immunogenitätsvorhersage
Die Immunogenitätsvorhersage (Schritt 4 in deinem Schema) bleibt eine der größten Herausforderungen, da sie die Interaktion zwischen MHC-Peptid-Komplex und T-Zell-Rezeptoren (TCR) betrifft. Neue Ansätze nutzen:
TCR-Sequenzdatenbanken: Große Datenbanken wie VDJdb oder McPAS-TCR enthalten Tausende von TCR-Sequenzen und deren bekannte Epitop-Bindungen. KI-Modelle trainieren auf diesen Daten, um Muster zu erkennen, die auf immunogene Epitope hinweisen.
Single-Cell-Sequenzierung: Diese Technologie ermöglicht es, die TCR-Repertoires einzelner T-Zellen zu analysieren. KI kann diese Daten nutzen, um patientenspezifische T-Zell-Reaktionen vorherzusagen, was die Personalisierung weiter verbessert.
Simulation von TCR-Epitop-Interaktionen: Fortschritte in der Molekulardynamik-Simulation, unterstützt durch KI, ermöglichen es, die physikalische Bindung zwischen TCR und MHC-Peptid-Komplex genauer zu modellieren.
4. Klinische Anwendungen
Die KI-gestützte Neoantigen-Identifizierung hat direkte Anwendungen in der personalisierten Medizin, insbesondere in:
Personalisierte Krebsimpfstoffe: Unternehmen wie BioNTech und Moderna nutzen KI-Pipelines, um Neoantigen-basierte mRNA-Impfstoffe zu entwickeln. Diese Impfstoffe kodieren für die vorhergesagten Neoantigene, um eine gezielte Immunantwort auszulösen. Klinische Studien (Stand 2025) zeigen vielversprechende Ergebnisse bei Melanom, Lungenkrebs und Pankreaskrebs.
Adoptive T-Zell-Therapien: KI hilft, Neoantigene zu identifizieren, die dann genutzt werden, um T-Zellen ex vivo zu trainieren (z. B. CAR-T-Zellen oder TCR-Therapien). Diese werden dem Patienten zurückgeführt, um den Tumor gezielt anzugreifen.
Kombinationstherapien: KI wird auch eingesetzt, um Neoantigene zu identifizieren, die mit Checkpoint-Inhibitoren (z. B. Anti-PD-1) kombiniert werden können, um die Immunantwort zu verstärken.
5. Herausforderungen und Grenzen
Neben den von dir erwähnten Herausforderungen (Datenqualität, Interpretierbarkeit, Immunediting) gibt es weitere Hürden:
Heterogenität der Tumore: Tumore sind oft heterogen, d. h., nicht alle Tumorzellen exprimieren die gleichen Neoantigene. KI-Modelle müssen lernen, Neoantigene zu priorisieren, die in der Mehrheit der Tumorzellen vorhanden sind.
Rechenaufwand: Obwohl KI den Prozess beschleunigt, sind die Berechnungen für die Integration von Multi-Omics-Daten und die Simulation komplexer molekularer Interaktionen immer noch rechenintensiv. Fortschritte in der Hardware (z. B. GPUs, TPUs) und Cloud-Computing helfen hier, aber es bleibt eine Herausforderung.
Klinische Validierung: Trotz der hohen Genauigkeit der KI-Vorhersagen bleibt die experimentelle Validierung (in vitro und in vivo) unverzichtbar. Die Diskrepanz zwischen vorhergesagten und tatsächlichen Immunantworten ist ein aktives Forschungsfeld.
6. Ausblick (Stand September 2025)
Open-Source-Tools und Kollaboration: Plattformen wie OpenCRAVAT oder NeoPredPipe machen KI-Tools für Neoantigen-Identifizierung für kleinere Forschungseinrichtungen zugänglich. Dies fördert die globale Zusammenarbeit und beschleunigt die Entwicklung.
Real-Time-Analyse: Fortschritte in der Sequenzierungstechnologie (z. B. Nanopore-Sequenzierung) und KI ermöglichen es, Neoantigene fast in Echtzeit während einer Operation oder Biopsie zu identifizieren.
Integration mit Wearables und Immunmonitoring: KI könnte in Zukunft mit Wearables kombiniert werden, um die Immunantwort nach einer Neoantigen-basierten Therapie in Echtzeit zu überwachen, was die Therapieanpassung ermöglicht.
Beispiel: Praktischer Workflow in der Klinik
Ein typischer Workflow in einer Klinik (2025) könnte so aussehen:
Tumor- und Blutprobe wird entnommen und sequenziert (DNA, RNA, ggf. Proteomik).
KI-Pipeline (z. B. eine Kombination aus NetMHCpan, MuPeXI und TCR-BERT) analysiert die Daten und liefert eine Liste von 10–20 priorisierten Neoantigen-Kandidaten.
Validierung: Die Top-Kandidaten werden in vitro getestet, um die Immunogenität zu bestätigen (z. B. durch ELISpot-Assays).
Therapieentwicklung: Die validierten Neoantigene werden in einen mRNA-Impfstoff oder eine T-Zell-Therapie integriert.
Klinische Anwendung: Der Patient erhält die personalisierte Therapie, oft kombiniert mit Checkpoint-Inhibitoren, gefolgt von Immunmonitoring.
Fazit
Die KI-gestützte Identifizierung von Neoantigen-Epitopen ist ein Paradebeispiel für die Verschmelzung von Bioinformatik, maschinellem Lernen und personalisierter Medizin. Sie hat das Potenzial, die Krebstherapie zu revolutionieren, indem sie präzise, patientenspezifische Immuntherapien ermöglicht. Fortschritte in Transformer-Modellen, Multi-Omics-Integration und TCR-Vorhersagen treiben das Feld weiter voran, aber Herausforderungen wie Tumorheterogenität und die Notwendigkeit experimenteller Validierung bleiben bestehen. Mit der zunehmenden Verfügbarkeit von Open-Source-Tools und der Integration von Echtzeit-Daten wird dieses Feld in den kommenden Jahren noch dynamischer werden.







MHCflurry ist ein hochspezialisiertes Bioinformatik-Tool und ein gutes Beispiel dafür, wie Künstliche Intelligenz (KI) und maschinelles Lernen in der immunologischen Forschung praktisch angewendet werden.
Kurz und knapp: Was ist MHCflurry?
MHCflurry ist ein open-source Software-Paket, das antizipiert, wie stark kurze Proteinabschnitte (Peptide) an MHC-I-Moleküle binden werden. Es nutzt dafür neuronale Netze (eine Form des maschinellen Lernens), die auf experimentellen Daten trainiert wurden.
Ausführliche Erklärung
Stellen Sie sich das vorhergesagte Peptid als einen Schlüssel und das MHC-Molekül als Schloss vor. MHCflurry sagt vorher, wie gut der Schlüssel ins Schloss passt.
1. Kontext: Warum ist das wichtig?
· Damit eine Krebszelle oder eine virusinfizierte Zelle vom Immunsystem erkannt wird, muss sie ein "fremdes" Peptid (z.B. von einem Virus oder einer Krebsmutation) auf…